目录准备数据简单热图只对列进行聚类将相关系数显示在图上在图上加上显著性标记准备数据 两组变量的数据可以像下面这样处理,分别保存在两个csv文件中。 > # 导入数据及数据预处理
两组变量的数据可以像下面这样处理,分别保存在两个csv文件中。
> # 导入数据及数据预处理
> setwd("D:/weixin/")
> rows <- read.csv("rows.csv")
> cols <- read.csv("cols.csv")
> str(rows)
'data.frame': 100 obs. of 6 variables:
$ r1: num 476 482 640 452 308 ...
$ r2: num 2059 1987 1952 1927 1854 ...
$ r3: num 513 601 682 497 463 ...
$ r4: num 2235 2114 2038 1945 1916 ...
$ r5: num 433 376 525 395 238 ...
$ r6: num 2028 1943 1802 1775 1748 ...
> str(cols)
'data.frame': 100 obs. of 5 variables:
$ c1: num 2387 2437 2484 2349 2198 ...
$ c2: num 540 535 706 509 359 ...
$ c3: num 472 610 465 473 471 ...
$ c4: num 74.4 57.3 49.5 51.8 47.6 ...
$ c5: num 995 915 1038 794 652 ...
> # 构建相关关系矩阵
> library(psych)
> data.corr <- corr.test(rows, cols, method="pearson", adjust="fdr")
> data.r <- data.corr$r # 相关系数
> data.p <- data.corr$p # p值
>
> # 画热图
> library(pheatmap)
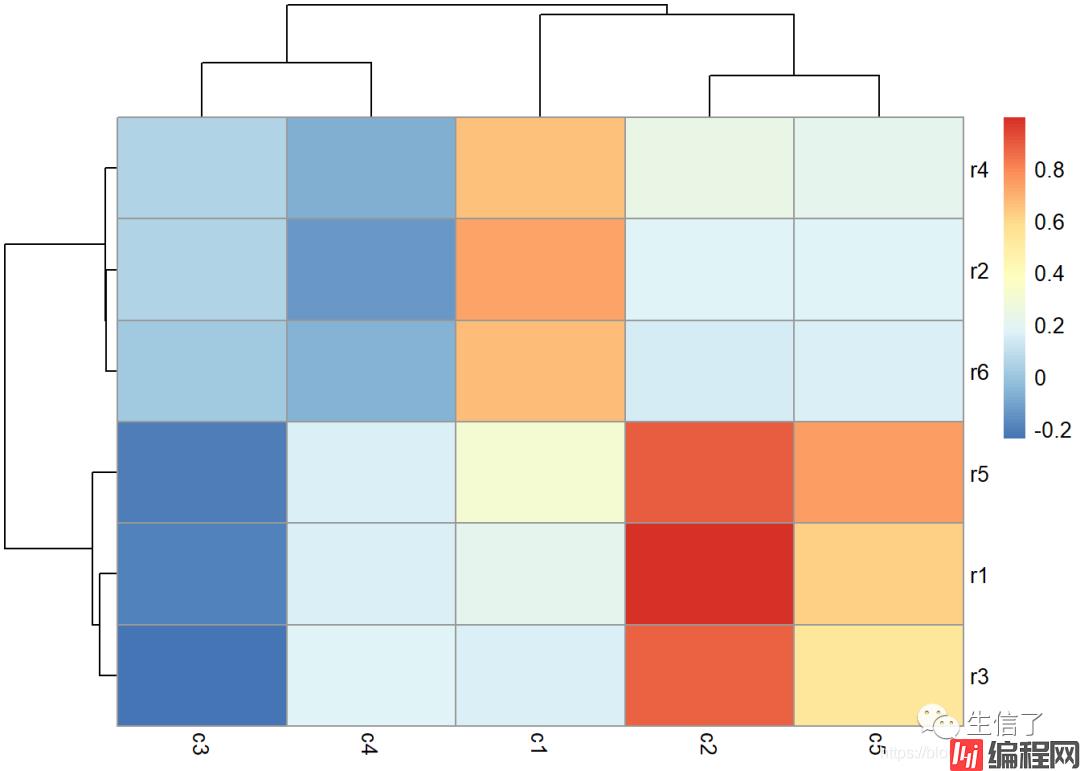
> pheatmap(data.r, clustering_method="average")
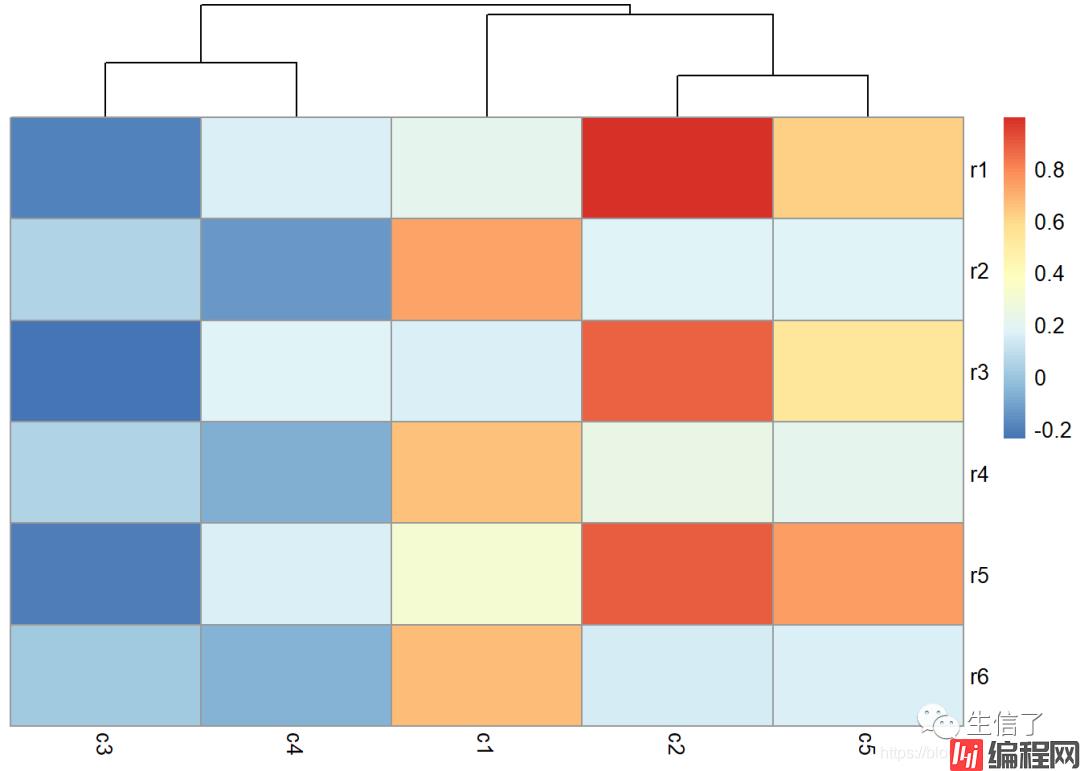
> pheatmap(data.r, clustering_method="average", cluster_rows=F)
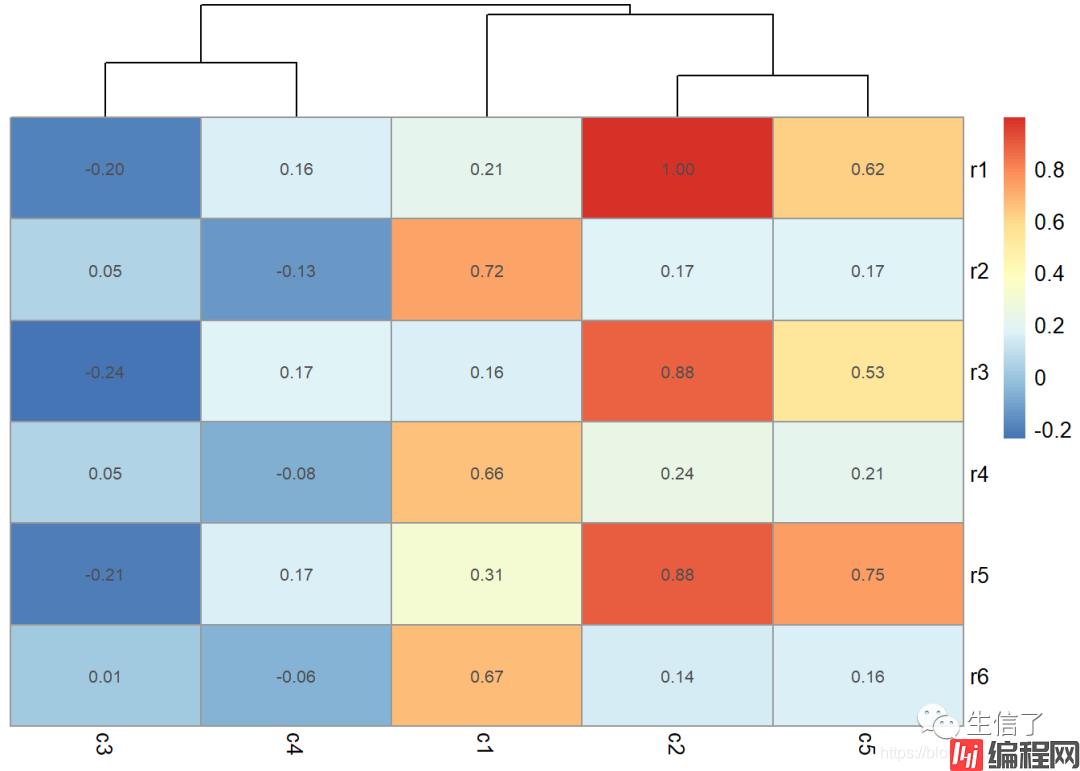
> data.r.fmt <- matrix(sprintf("%.2f", data.r), nrow=nrow(data.p)) # 只保留小数点后两位
> pheatmap(data.r, clustering_method="average", cluster_rows=F, display_numbers=data.r.fmt)
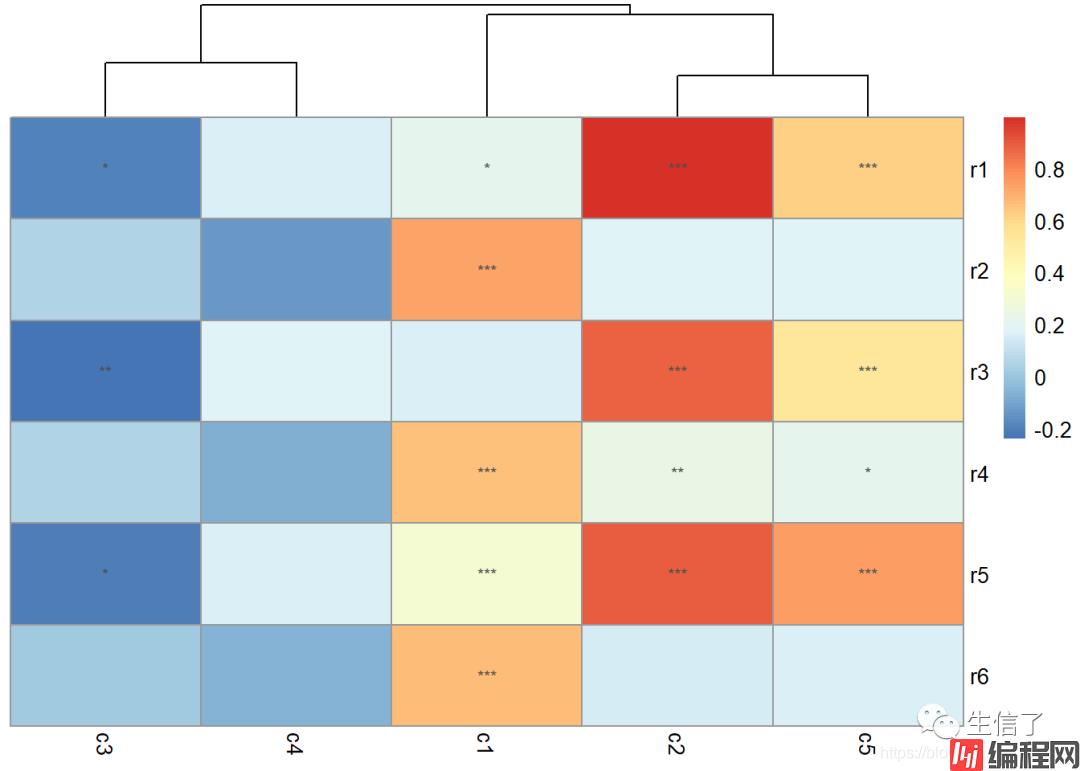
> getSig <- function(dc) {
+ sc <- ''
+ if (dc < 0.01) sc <- '***'
+ else if (dc < 0.05) sc <- '**'
+ else if (dc < 0.1) sc <- '*'
+ sc
+ }
> sig.mat <- matrix(sapply(data.p, getSig), nrow=nrow(data.p))
> str(sig.mat)
chr [1:6, 1:5] "*" "***" "" "***" "***" "***" "***" "" "***" "**" ...
> pheatmap(data.r, clustering_method="average", cluster_rows=F, display_numbers=sig.mat)
如果想进一步改变图形效果,可以参考pheatmap函数的用法,修改相应的参数。比如:聚类方式改为complete,加上标题等。
> pheatmap(data.r, clustering_method="complete", cluster_rows=F, display_numbers=sig.mat, main="Corr Heatmap")
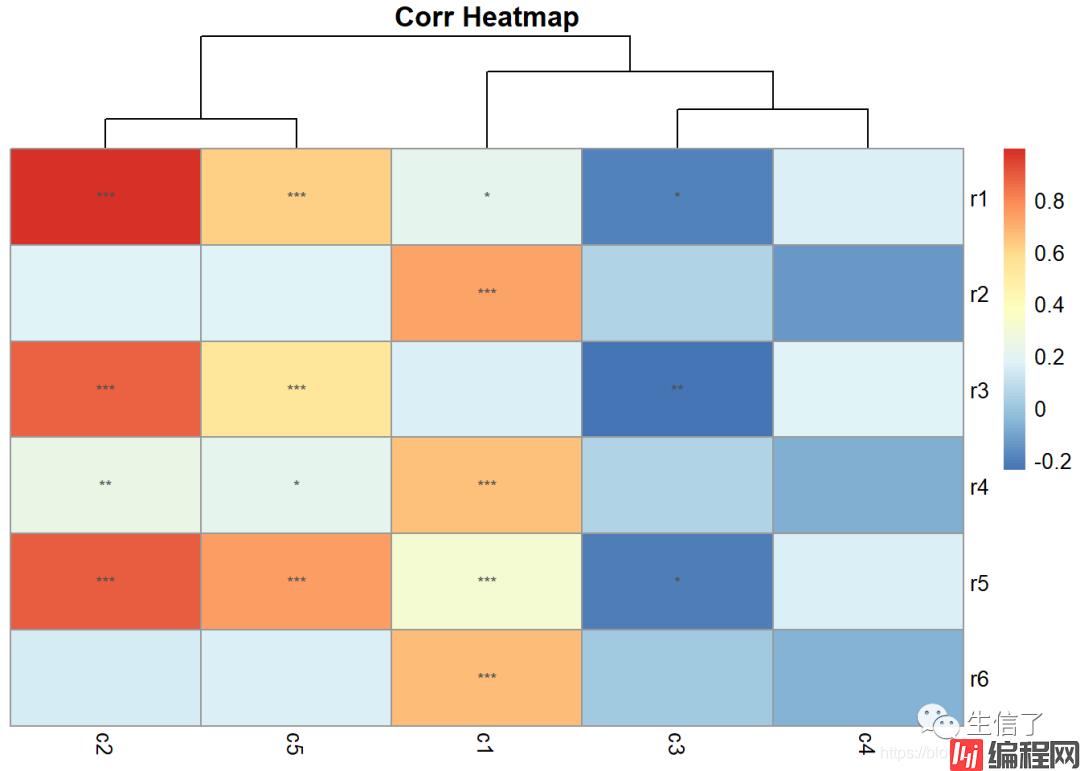
以上就是R语言两组变量特征相关关系热图绘制画法的详细内容,更多关于R语言绘制相关关系热图的资料请关注编程网其它相关文章!
--结束END--
本文标题: R语言两组变量特征相关关系热图绘制画法
本文链接: https://lsjlt.com/news/139609.html(转载时请注明来源链接)
有问题或投稿请发送至: 邮箱/279061341@qq.com QQ/279061341
2024-03-01
2024-03-01
2024-02-29
2024-02-29
2024-02-29
2024-02-29
2024-02-29
2024-02-29
2024-02-29
2024-02-29
回答
回答
回答
回答
回答
回答
回答
回答
回答
回答
0